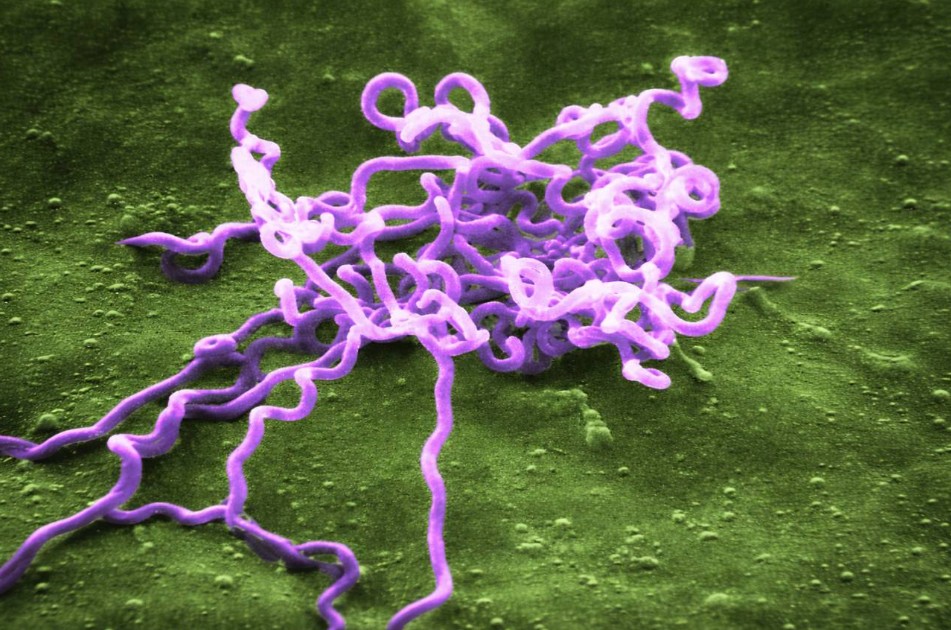
Un equipo internacional de científicos halló ADN de la bacteria Treponema pallidum —causante de la sífilis— en los restos de un cazador que vivió hace 5 500 años en lo que hoy es Colombia. El descubrimiento constituye el registro más antiguo conocido de este patógeno, responsable de una de las infecciones más devastadoras de la historia.
Por: Federico Kukso
Hace 5 500 años, un cazador murió a unos 30 kilómetros al suroeste de lo que hoy es Bogotá, Colombia. No era alto —apenas alcanzaba el metro sesenta— y tenía algo más de 45 años, una edad considerable para su tiempo. Siguiendo los ritos de su gente, sus familiares acomodaron su cuerpo en un refugio rocoso que, siglos después, sería llamado Tequendama I.
Nadie sabe cómo se llamaba este hombre, cómo sonaba su voz ni la causa exacta de su muerte. Lo único que los científicos han podido confirmar, tras un análisis paleogenético minucioso, es que durante todo ese tiempo su esqueleto guardó un secreto. “Para nuestra sorpresa, en un hueso de la pierna encontramos ADN de la bacteria Treponema pallidum, responsable de varias enfermedades como la sífilis, el pian y el bejel”, cuenta a SINC el arqueólogo colombiano y uno de los autores de la investigación Miguel Delgado.

En un estudio publicado en la revista Science, este investigador de la Universidad Nacional de La Plata (Argentina) y un equipo de genetistas italianos, estadounidenses, alemanes y suizos revelan que se trata de la evidencia molecular más antigua conocida a escala global de este patógeno en humanos. El hallazgo redefine lo que se sabía hasta ahora sobre una de las enfermedades infecciosas que más ha afectado a las poblaciones en el mundo.
“Conocer la historia evolutiva de esta enfermedad nos ayuda a comprenderla en profundidad”, agrega Delgado, conocido por su estudio del poblamiento americano. “Nos permite entender un poco mejor su manifestación y su dispersión actual”.
Un hallazgo con suerte
El sitio arqueológico Tequendama I se ubica en la Sabana de Bogotá en las tierras altas orientales de Colombia, una región que jugó un papel clave en la expansión humana inicial en América del Sur. Desde 1969, arqueólogos como Gonzalo Correal Urrego han recuperado allí herramientas líticas, arte rupestre, huesos de animales y cientos de restos de humanos bien conservados, algunos con una antigüedad de hasta 12 mil años. Entre ellos, se encuentra el cuerpo de este hombre de 1,58 metros de altura, identificado como ‘TE1-3’.

Ubicación del sitio arqueológico Tequendama en la región de Sabana de Bogotá en Colombia./ Instituto Colombiano de Antropología e Historia (ICANH).
El análisis de radiocarbono indica que vivió hace aproximadamente 5 500 años. No presenta signos claros sobre la causa de su muerte: no hay evidencia de violencia ni de enfermedades complejas. “Probablemente se trató de una muerte natural, consecuencia de un estilo de vida extremadamente duro y de una alimentación precaria”, sospecha el arqueólogo.
El hallazgo del ADN de esta bacteria fue, en cierto modo, fortuito, explican los científicos. Para reconstruir la historia evolutiva de las poblaciones y las enfermedades que padecieron, los paleogenetistas suelen examinar ADN conservado en el oído interno o en dientes, donde se preserva la mayor cantidad de material genético prehistórico. “Sin embargo, a la hora de tomar las muestras no contábamos ni con dientes ni con el cráneo”, advierte Delgado, científico del Conicet en Argentina.
Tras obtener una licencia del Instituto Colombiano de Antropología e Historia (ICANH) —el ente regulador del patrimonio arqueológico en Colombia—, los investigadores decidieron asumir el riesgo y enviaron muestras de la tibia de este cazador a distintos laboratorios para realizar análisis de ADN antiguo.
Primero, al Laboratorio de Paleogenómica Humana del antropólogo biológico alemán Lars Fehren-Schmitz, en la Universidad de California, Estados Unidos. Luego al Grupo de Genómica de Poblaciones de la paleogenetista Anna-Sapfo Malaspinas, en la Universidad de Lausana, Suiza. Al secuenciar el material, los científicos advirtieron que allí había un tipo de ADN que no pertenecía al individuo, sino a una bacteria con forma helicoidal: Treponema pallidum.

“Fue realmente sorprendente”, asegura la antropóloga molecular Elizabeth A. Nelson, de la Universidad Metodista del Sur en Dallas y una de las autoras del artículo. “Recuperar ADN de patógenos antiguos siempre es un desafío, y el Treponema pallidum es particularmente difícil de detectar. Esto se debe a que, durante las etapas de formación de lesiones esqueléticas, el patógeno suele estar presente en el cuerpo en niveles muy bajos”.
Este descubrimiento extiende la compleja historia evolutiva de este patógeno: es tres mil años más antiguo que los restos humanos con T. pallidum hallados en la costa sureste de Brasil —reportados en 2024— y cinco mil años anterior a la llegada de los europeos al continente.
La biografía secreta de los patógenos
El origen geográfico, la evolución y la propagación de las “enfermedades treponémicas” —cada una causada por subespecies de la diversa familia de microbios Treponema— constituyen uno de los debates más persistentes en la historia de las enfermedades infecciosas. Sin tratamiento, la sífilis puede provocar consecuencias neurológicas devastadoras: ceguera, sordera, trastornos mentales e incluso la muerte.
Es, además, una de las infecciones de transmisión sexual que mayor preocupación suscita: según la Organización Mundial de la Salud, su incidencia global aumenta año tras año. La frambesia (o pian), en cambio, afecta sobre todo a la piel y los huesos, y se transmite por contacto directo en regiones cálidas y húmedas. El bejel y el mal de pinto, por su parte, producen úlceras bucales y lesiones óseas.
Dónde y cuándo aparecieron por primera vez los patógenos que han afectado a las sociedades humanas sigue siendo un gran misterio. Los recientes avances tecnológicos y la reducción de los costes de secuenciación han permitido el estudio sin precedentes de la evolución de bacterias, virus y parásitos.
En especial, la investigación del ADN antiguo (aDNA) de patógenos proporciona información única sobre infecciones microbianas pasadas. Este material hereditario funciona como una cápsula del tiempo: posee una estabilidad notable, puede analizarse incluso después de milenios y permite observar cómo los patógenos han cambiado genéticamente con el tiempo.
“El campo del ADN antiguo despegó con la llegada de la secuenciación de alto rendimiento en 2008”, recuerda el biólogo molecular argentino Nicolás Rascován, del Instituto Pasteur en París. “En 2011, se recuperó por primera vez el genoma de un patógeno antiguo a partir de muestras arqueológicas, el de la bacteria Yersinia pestis, responsable de la peste. Fue entonces cuando tomó fuerzas el estudio de enfermedades del pasado a través de la secuenciación de ADN”.
Desde ese momento, la floreciente ciencia de la ‘paleogenómica’ permitió reconstruir la historia profunda de los patógenos responsables de enfermedades infecciosas humanas, como la tuberculosis (Mycobacterium tuberculosis), la viruela (el virus Variola) y la hepatitis B (VHB). Diversos estudios muestran que estas infecciones emergieron hace unos 6 500 años, en coincidencia con el inicio de la convivencia estrecha entre humanos y animales domesticados, un cambio que incrementó el riesgo de zoonosis, es decir, de la transmisión de patógenos de animales a personas.
“Al estudiar el ADN de patógenos antiguos, podemos rastrear su evolución a medida que se desarrollaban”, explica el biólogo computacional italiano Davide Bozzi, de la Universidad de Lausana. “Esto puede brindarnos información sobre cómo los patógenos se adaptaron para infectar a los humanos o cómo se volvieron virulentos. Este conocimiento es un primer paso esencial para comprender y controlar la aparición de nuevos virus y bacterias o la reaparición de enfermedades pasadas”.
Un enemigo invisible
En la primavera de 1495, el rey francés Carlos VIII invadió el reino de Nápoles, impulsado por el deseo de convertirse en emperador. Tras un breve éxito, sus tropas de mercenarios empezaron a sucumbir en manos de un enemigo invisible, una enfermedad desfigurante hasta entonces desconocida. Las crónicas médicas de la época describen fiebre, erupciones cutáneas generalizadas, úlceras genitales, dolores óseos intensos y una elevada mortalidad.
Los franceses la llamaron “la enfermedad napolitana”. Para los italianos era “morbus gallicus” (enfermedad francesa). Los rusos la conocían como “la enfermedad polaca” y los polacos, “la enfermedad alemana”. Para los habitantes de Flandes y el norte de África era “la enfermedad española”, mientras que para los españoles era “las bubas”.

Izq.: Un hombre sifilítico, xilografía atribuida a Albrecht Dürer, 1496. Der.: Página de un tratado temprano sobre la sífilis, 1495-1498. / Colección Wellcome.
En pocos meses, el mal se propagó por Europa siguiendo las rutas militares y comerciales, dejando a muchos sobrevivientes con secuelas neurológicas y deformaciones permanentes. Hoy, ese episodio se interpreta como el primer registro histórico documentado de la sífilis en el Viejo Mundo.
Algunos creyeron que se trataba de un castigo divino. Para los astrólogos, la epidemia era consecuencia de dos eclipses de sol y de la confluencia de Marte y Saturno. Otros, en cambio, culparon a los marineros que regresaron de América con Cristóbal Colón en 1493. “Es posible que la sífilis del siglo XV fuera la primera enfermedad infecciosa emergente globalizada y un presagio de todas las posteriores, desde el VIH/sida hasta la COVID-19”, afirman las antropólogas Molly K. Zuckerman y Lydia Bailey de la Universidad Estatal de Mississippi.
El término sífilis fue acuñado recién en 1530 por el médico y poeta italiano Girolamo Fracastoro, en su poema Syphilis sive Morbus Gallicus («Sífilis o la enfermedad francesa»). Allí, el pastor Syphilius ofende al dios Apolo y, como castigo, desata una plaga que azota a su pueblo. La enfermedad recibe su nombre.
Desde entonces hay varias hipótesis en pugna que buscan explicar su origen. La “hipótesis colombina” sostiene que la sífilis ya estaba extendida en las poblaciones humanas del continente americano antes de la conquista europea. La “hipótesis precolombina”, en cambio, afirma que la bacteria T. pallidum circulaba en Eurasia en el período medieval y posiblemente antes, pero era confundida con otras enfermedades. El consenso actual es que se encontraba presente en ambos continentes, pero en América una variante habría evolucionado hacia la sífilis venérea. El contacto europeo habría acelerado su rápida propagación global.
“La pregunta por el origen de la enfermedad se ha planteado durante siglos porque la gente quería culpar a alguien”, apunta la antropóloga Nasreen Broomandkhoshbacht, de la Universidad de California. “Estas narrativas son dañinas; pueden usarse para justificar la violencia y la deshumanización de grupos enteros de personas”.
Pistas sobre la evolución de la enfermedad
Desde su descubrimiento en 1905, esta bacteria ha intrigado a los microbiólogos, en especial por la imposibilidad de cultivarla en el laboratorio. En 1943, la penicilina revolucionó el tratamiento de la sífilis. En 1998 su genoma completo fue secuenciado. Por entonces, se pensaba que ese logro iba a allanar el camino para el desarrollo de vacunas preventivas. Eso no ha ocurrido.
El ADN antiguo de T. pallidum, identificado en la tibia de este cazador-recolector que vivió hace 5 500 años en Colombia, aporta claves valiosas para comprender la evolución de la enfermedad. Al comparar este material genético con el de cepas modernas del mismo patógeno, es posible rastrear los cambios que ha experimentado a lo largo del tiempo. En este caso, el análisis filogenético revela que el genoma de esta bacteria corresponde a una rama hasta ahora desconocida de Treponema pallidum, que divergió antes del ancestro común de las subespecies actuales.
Aunque se sitúa claramente dentro de la especie T. pallidum, la cepa TE1-3 presenta una diversidad genética notable y es distinta de las cepas modernas. “Lo que encontramos no puede clasificarse como sífilis”, advierte Broomandkhoshbacht.
“La sífilis, el bejel y el pian son enfermedades muy diferentes, y todas están mucho más relacionadas entre sí que con el patógeno que describimos en nuestra investigación. Ahora sabemos que en el pasado existía una diversidad aún mayor de estos patógenos, lo que podría significar que había una variación aún mayor en su comportamiento. Los registros históricos de la sífilis muestran que ha cambiado drásticamente en los últimos 500 años, por lo que incluso si lo que encontramos fuera un ancestro directo de la sífilis (que no lo es), podríamos asumir que actuó de manera bastante diferente”, añade.
Los hallazgos sugieren, además, que T. pallidum es anterior al surgimiento de la agricultura en el continente americano. “Esta persona vivió hace unos 5 500 años, en una comunidad de cazadores-recolectores del Holoceno Medio, mucho antes de la intensificación agrícola o los cambios demográficos de la época colonial, dos factores que a menudo se asocian con la propagación de enfermedades infecciosas”, agrega Nelson.
Condiciones de los cazadores-recolectores
En cambio, el linaje TE1-3 parece vincularse a las condiciones sociales y ecológicas propias de las sociedades de cazadores-recolectores: alta movilidad, interacciones en comunidades pequeñas y un estrecho contacto con animales salvajes.
Aun así, a los científicos les resulta llamativo que los restos del individuo estudiado no presenten signos visibles de enfermedad. “No podemos determinar si la ausencia de patología esquelética refleja una fase temprana o latente de la infección”, afirman.
Todavía quedan muchas preguntas sin resolver. “Aún desconocemos cómo se transmitió este antiguo linaje, si los animales desempeñaron un papel como reservorios en el pasado o si el patógeno que identificamos representa un linaje temprano o ya extinto”, enumera Elizabeth Nelson.
Además de continuar sus investigaciones en Colombia, el equipo científico está ampliando el alcance de su trabajo hacia el sur, en los Andes, en colaboración con colegas peruanos. “Para comprender cómo evolucionó el linaje de Treponema identificado en Tequendama, es fundamental estudiar a otros individuos a lo largo del tiempo y en distintas regiones”, señala el antropólogo Lars Fehren-Schmitz. “Asimismo, para entender mejor la ecología de las enfermedades, la investigación no debe limitarse a los restos de ancestros humanos, sino extenderse también al análisis de restos animales”.
Referencia: Miguel Delgado et al. A 5,500-year-old Treponema pallidum genome from Sabana de Bogotá, Colombia. Science (2026).